Lesson 5 Prefix Sums - GenomicRangeQuery (Java Solution)
app.codility.com/programmers/lessons/5-prefix_sums/genomic_range_query/
GenomicRangeQuery coding task - Learn to Code - Codility
Find the minimal nucleotide from a range of sequence DNA.
app.codility.com
문제
DNA sequence는 A, C, G, T로 구성되어 있다. 각각 1,2,3,4에 영향을 가진다
S = S[0]S[1]...S[N-1], M은 P와 Q의 배열 사이즈이다
K-th query (0<= K < M)는 P[K]와 Q[K] 사이 DNA sequence에 포함된 nucleotides의 최소의 영향 계수를 찾는 것이다
예를 들면,
S = CAGCCTA 이고, P = {2,5,0} , Q = {4,5,6}, M=3이다
1) P[0] = 2 , Q[0] = 4 -> S[2]S[3]S[4] : GCC => 3과 2이므로 minimal impact는 2이다
2) P[1] = 5, Q[1] = 5 -> S[5] : T => 4이므로 minimal impact는 4이다
3) P[2] = 0, Q[2] = 6 -> S[0]S[1]~S[6] = CAGCCTA => 1,2,3,4이므로 minimal impact는 1이다
즉 결과는 [2,4,1]이다
해결
문제 그대로 S, P, Q를 정의하고,
A,C,G,T를 static Map에 1,2,3,4,로 대응시켜두었다
그리고 M만큼 반복하면서 최소값을 구해서 minArr에 넣었다
public class GenomicRangeQuery {
public final static Map<Character, Integer> sequenceMap = new HashMap<>() {
{
put('A', 1);
put('C', 2);
put('G', 3);
put('T', 4);
}
};
public int[] solution(String S, int[] P, int[] Q) {
int M = P.length;
char[] charArray = S.toCharArray();
int[] minArr = new int[M];
for (int i=0; i<M; i++) {
List<Integer> list = new ArrayList<>();
for (int j=P[i]; j<=Q[i]; j++) list.add(sequenceMap.get(charArray[j]));
minArr[i] = list.stream().min(Integer::compareTo).get();
}
return minArr;
}
}테스트 케이스는 모두 통과했지만, 성능 테스트는 0%가 나왔다
현재 시간복잡도는 O(N*M)이다
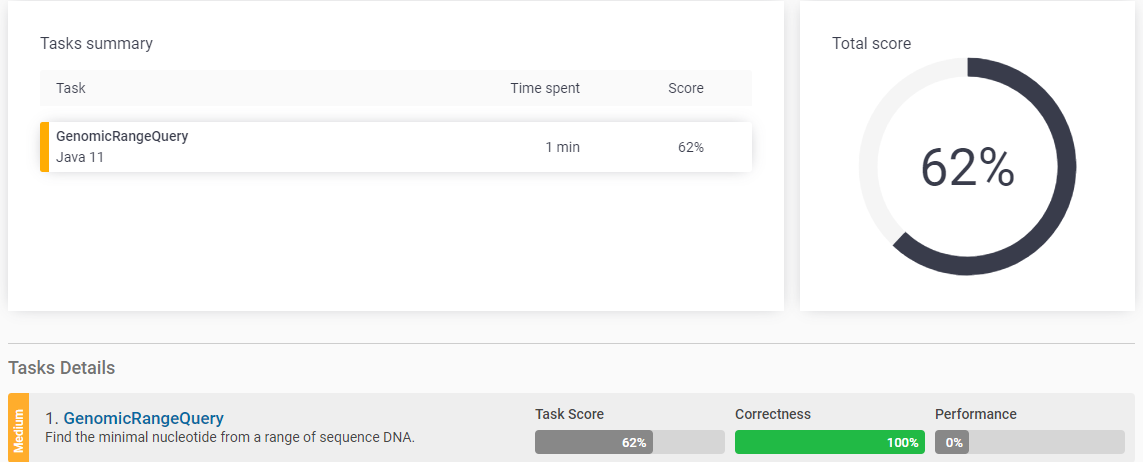
문제는 어렵지 않지만, 시간 복잡도를 해결하기가 어려웠다
검색해보니 O(N+M)이하의 시간 복잡도가 나와야 성능 테스트의 100%가 나온다고 하여 다시 풀어보았다
우선, O(N+M)이 나오려면 for 문안에서 최솟값을 계산하면 안 된다
단어를 카운트하는 방식으로 바꿨다
먼저 Map에 A,C,G,T Index 값을 정의한다 (A :0, C:1, G:2, T:3)
S = CAGCCTA을 하나씩 돌면서 Count 값을 올린다
예를 들면,
C : {0,1,0,0}
CA : {1,1,0,0}
CAG : {1,1,1,0}
CAGC : {1,2,1,0}
CAGCC: {1,3,1,0}
그리고 2차원 배열에 [[0100],[1100],[1110],[1210]...] 으로 쌓는다
이제 답을 찾아보자
P = {2,5,0} , Q = {4,5,6}, M=3이다
1) P[0] = 2 , Q[0] = 4 -> 위에서 쌓은 2차원 배열의 4번째 Index에서 (2-1)번째 Index를 뺀다
-> {1,3,1,0} - {1,1,0,0} = {0,2,1,0} -> 위에서 찾은 GCC와 같은 값이다
minimal impact 값을 찾으려면 앞에서부터 순회하여 0보다 큰 index+1을 하면 리턴하면 결과값이 나온다
index+1을 하는 이유는 A,C,G,T를 매핑할 때 배열의 인덱스 때문에 1씩 낮게 매핑해뒀기 때문이다
public class GenomicRangeQuery {
public final static Map<Character, Integer> sequenceMap = new HashMap<>() {
{
put('A', 0);
put('C', 1);
put('G', 2);
put('T', 3);
}
};
public int[] solution(String S, int[] P, int[] Q) {
char[] charArray = S.toCharArray();
int[] count = {0,0,0,0};
int[][] countArry = new int[charArray.length][count.length];
for (int i=0; i<charArray.length; i++) {
count[sequenceMap.get(charArray[i])]++; // [0100], [1100], [1110], [1210] ....
for (int j=0; j<count.length; j++) {
countArry[i][j] = count[j]; // [[0100],[1100],[1110],[1210]....]
}
}
int M = P.length;
int[] minArr = new int[M];
for (int i=0; i<M; i++) {
for (int j=0; j<count.length; j++) {
int end = countArry[Q[i]][j];
int before_start = P[i] == 0 ? 0 : countArry[P[i]-1][j];
if (end - before_start > 0) {
minArr[i] = j+1;
break;
}
}
}
return minArr;
}
}
'Algorithm > Codility' 카테고리의 다른 글
| [Codility(코딜리티)] Lesson 13. FibFrog (100%) (0) | 2020.09.30 |
|---|---|
| [Codility(코딜리티)] Lesson7. Nesting (100%) (0) | 2020.09.14 |
| [Codility(코딜리티)] Lesson7. Fish (100%) (0) | 2020.09.13 |
| [Codility(코딜리티)] Lesson5. CountDiv (100%) (0) | 2020.09.12 |
| [Codility(코딜리티)] Lesson4. PermCheck (100%) (0) | 2020.09.12 |




댓글